서 론
24시간을 주기로 자전하는 지구상의 모든 유기체는 하루 중 시간에 따라 변화하는 환경 신호에 맞춰 유기체 내부의 생물학적 과정을 조정하는 일주기 시계를 갖게 되었다[1]. 일주기 시계는 유기체의 생리작용과 발달 과정의 생물학적 리듬을 생성하는 분자 타이밍 장치이다[2]. 일일주기 리듬을 조절하는 생체시계(circadian clock) 유전자는 식물 생명의 주된 조절자 역할을 하며, 환경 적응 반응을 촉진하는 다양한 신호 및 대사 경로와 연결되어 있다. 환경과 식물 내생리듬의 올바른 일치는 적응력 향상에 기여하여 생존과 최적의 생장과 발달을 보장한다. 일주기 리듬(circadian rhythm)은 내생적으로 생성되고 자체적으로 유지되며, 다양한 생리적 온도에서 견고하고 정확한 타이밍을 유지할 수 있다[3,4].
온도는 작물의 품질과 생산성뿐만 아니라, 식물의 생장과 발달 및 지리적 분포에 영향을 미친다. 온도에 의존하는 식물 반응에는 열주기(thermoperiodism), 열형태형성(thermomorphogenesis), 저온성층화(cold strafication), 극한 온도 반응(extream-temperature responses) 등이 포함된다. 하루를 주기로 변화하는 온도는 식물체에 시간 정보를 제공하여 리듬을 만드는 신호로 작용(entrainment)하는 것으로 알려져 있지만, 시계 주기는 생리학적으로 관련된 온도 범위에서 크게 변하지 않고 일정하게 유지될 수 있는 기능을 가지며, 이를 온도보상(temperature compensation) 이라고 한다[5]. 온도보상뿐만 아니라 극단적인 온도 변화 또는 식물의 생장과 발달에 영향을 주는 스트레스 온도 조건에서도 생체리듬을 안정화시키는 기작들이 연구되고 있다. 저온 조건에서 CCA1 (CIRCADIAN CLOCK ASSOCIATED 1) 유전자의 대체스플라이싱(alternative splicing)이 일어난다거나, E3 ubiquitin ligase인 ZTL(ZEITLUPE)와 HSP90(HEAT SHOCK PROTEIN 90)이 고온 조건에서 식물 생체시계를 안정화시키는 독특한 메커니즘을 가진다는 보고가 그 대표적인 예이다[6]. 세포 대사의 변화에 대응하여 리듬을 조절하는 분자 메커니즘은 다양한 식물 종에서 탐구되고 있으며, 시계 주기의 안정화는 식물의 정상적인 생장과 발달을 유지하고, 불리한 환경 조건에서의 생존을 가능하게 한다.
식물이 견딜 수 있는 적정한 범위의 온도 변화나 스트레스가 되는 극단적 온도 변화에 대한 식물의 적응 메커니즘에 대한 연구는 식물이 주변 환경 변화에 적응하여 생산성을 유지할 수 있는 중요한 기초적인 자료를 제공할 수 있으며, 특히 지구 온난화에 의해 작물의 재배지가 위협을 받는 현 시점에서 미래 식량 유지를 위한 육종 소재를 제공할 수 있다. 본 리뷰에서는 온도 변화에 대한 반응 기작과 스트레스 온도에 적응하는데에 관여하는 생체리듬 조절 유전자들과 기작을 소개하고자 한다.
본 론
식물 시계 시스템은 여러 개의 억제 피드백 루프를 포함하는 복잡한 유전자 회로로 알려져 있다[7]. 생체리듬을 조절하는 전사 조절 네트워크 모델 시스템은 애기장대(Arabidopsis thaliana)에서 많이 연구되었다. 중앙진동자(main oscillator)의 피드백 루프는 CCA1, LHY (LATE ELONGATED HYPOCOTYL), TOC1(TIMING OF CAB EXPRESSION 1)[PRR1(PSEUDO-RESPONSE REGULATOR1)이라고도 함] 및 MYB 전사인자인 LUX (LUX ARRHYTHMO ), 그리고 추가적인 전사조절인자 ELF3(EARLY FLOWERING 3 ) 및 ELF4 를 포함하는 EC (EVENING COMPLEX )로 구성된다. 이 중앙진동자 피드백 루프의 CCA1 및 LHY 발현은 아침에 최고조에 달하면서 TOC1 유전자의 전사를 억제하는 반면, TOC1 발현은 저녁 시간에 최고조에 달하고, CCA1 및 LHY 유전자의 전사를 억제한다. 또한, CCA1와 LHY 는 PRR7 및 PRR9 유전자를 활성화하고, 이 유전자는 시간 순서에 따라 차례로 CCA1 유전자 전사를 억제한다. 한편, TOC1은 EC 구성요소, PRRs 및 GI (GIGANTEA) 유전자의 전사를 억제한다. 중앙진동자의 다른 주요 구성 요소에는 GI, ZTL 및 PRR3이 포함된다. LHY 와 CCA1은 이른 아침에 GI 전사를 억제하는 반면, GI 전사는 한낮에 정점에 도달한다[8,9].
청색광 수용체이면서 TOC1 분해를 매개하는 E3 ubiquitin ligase로서 중요한 일주기 조절 시스템 단백질인 ZTL의 발현은 황혼에 최고조에 이르며, GI 는 LOV(light, oxygen, voltage) 도메인을 통해 ZTL과 상호 작용한다[10]. 이 단백질-단백질 상호 작용은 청색광에서 ZTL 단백질을 안정화시켜 ZTL의 축적을 유도하고, GI 가 HSP90의 샤페론 활동과 함께 낮 동안 ZTL의 성숙을 강화한다는 것이 증명되었다[11]. 일주기 리듬을 조절하는 시계 유전자들의 돌연변이는 중앙진동자의 기능에 영향을 미쳐 일주기 리듬의 주기와 진폭을 변화시킬 뿐 아니라 다양한 환경 스트레스 조건에 대한 식물의 반응과 적응 능력을 변경한다. prr5/7/9 삼중 돌연변이는 고염도와 가뭄 스트레스에 더 잘 견딘다[12]. GI 과발현 식물은 향상된 염분 민감성을 보이는 반면, gi 돌연변이체는 가뭄 스트레스 및 산화 스트레스 하에서 향상된 염분 내성 및 향상된 생존을 보인다[13,14]. TOC1-RNAi 식물에서 TOC1의 발현 감소는 가뭄 내성을 개선하는 반면, TOC1의 과발현은 가뭄 조건에서 수분 손실을 증가시키고 생존율을 감소시킨다[15]. prr5/7/9 및 toc1 돌연변이체는 상당히 증가된 동결 내성을 나타내지만[12,16], gi, lux, lhy, cca1 및 lhy×cca1 돌연변이체는 동결 내성을 감소시켰다[17,18]. 이러한 발견은 일주기 시계가 환경 스트레스를 견디는 식물의 능력에 기여한다는 것을 보여준다.
주위 환경의 변화에 맞춰 정확한 타이밍에 생체시스템을 가동하고 유지하는 것은 식물의 생존과 최적의 생장에 필수적이다. 일주기 시계는 식물이 일주기 리듬을 유지할 수 있도록 하는 분자 완충 시스템을 가지고 있어 시계 출력 경로가 온도 변동의 생리적 범위에 크게 둔감하도록 만드는 것으로 알려져 있다. 이 현상을 온도 보상이라고 한다[5]. 온도 보상 메커니즘은 다양한 식물종에서 잘 보존되어 있으며, 일주기 시계 시스템의 중요한 속성이다. 시계 유전자는 아니지만, 부분적으로 열 감지기의 역할을 수행하는 phyB(PHYTOCHROME B)가 시계에 온도 정보를 전달할 수 있으며[19,20], PRR7 및 PRR9가 결여된 애기장대 식물이 온난-냉간 주기에 반응하여 일주기 리듬을 유지할 수 없음이 보고되었으며, PRR 단백질도 시계의 온도 반응에 있어 중요한 역할을 하는 것이 증명되었다[21]. 이외에도 FBH1(FLOWERING BASIC HELIX-LOOP-HELIX 1)과 HSF(HEAT SHOCK TRANSCRIPTION FACTOR) 단백질 그룹도 고온에 대한 식물 생체시계의 반응에 중요한 역할을 한다[22]. 한편, FBH1의 과잉 생산은 온도 변화에 대한 CCA1 반응을 조절함으로써 시계의 속도를 변경하는 것으로 밝혀졌다. 열 반응성 HsfB2b 전사인자는 온도가 상승하면, PRR7 전사를 억제하고, HsfB2b 발현이 억제되면 동일한 온도 조건에서 일주기가 짧아진다[23]. 이러한 결과들은 생체시계 유전자가 온도 신호를 전달하는 기능을 가질 가능성을 시사하며, 다른 비생물학적 스트레스와 같이 온도 스트레스에 대한 저항성에 영향을 줄 수 있음을 제시하였다.
애기장대에서 DREB1/CBFs(DEHYDRATION-RESPONSE ELEMENT-BINDING PROTEIN 1/C-REPEAT BINDING FACTOR)는 저온 반응성 유전자 발현에 관여하는 주요 전사인자로 확인되었다[24,25]. 이러한 DREB1/CBFs는 AP2/ERF(APETALA2/ETHYLENE-RESPONSIVE FACTOR) 전사인자로, DRE(DEHYDRATION-RESPONSE ELEMENT)에 결합하고 저온 유도 유전자 발현에서 마스터 조절 인자 역할을 한다. 애기장대에는 DREB1A/CBF3, DREB1B/CBF1 및 DREB1C/CBF2의 세 가지 DREB1/CBF 유전자가 있다. 이 유전자들은 애기장대 게놈에서 DREB1B, DREB1A 및 DREB1C 순서로 나란히 배열되어 있는데, 3개의 유전자 모두가 파괴된 애기장대 돌연변이 식물은 심하게 손상된 동결 내성을 보인다. 세 가지 유전자 모두 저온 스트레스에 의해 빠르게 유도되며, DREB1 유전자의 유도는 저온 반응성 전사 캐스케이드를 촉발하고, 이어서 다수의 저온 유도성 유전자의 발현을 유발한다. 따라서 저온 스트레스에 대한 DREB1 발현 메커니즘에 대한 연구는 식물의 저온 스트레스에 대한 인식과 반응을 이해하는데 중요하다. DREB1 유전자 자체는 저온 스트레스에 의해 빠르게 유도된다.
DREB1A를 포함한 많은 저온 유발 유전자의 발현은 정상 온도에서는 정오쯤에 발현하지만 발현 수준이 매우 낮다[26,27]. 24시간 주기 시계의 중앙 진동자의 핵심 구성 요소인 PRR9, PRR7 및 PRR5를 암호화하는 유전자의 돌연변이는 DREB1 유전자 및 그 하위 유전자의 발현을 향상시킨다[12,28]. 또한, DREB1 유전자의 저온 유도 발현은 CCA1과 LHY의 이중돌연변이 식물에서 유의하게 감소되었다. 이러한 결과는 DREB1A를 포함한 많은 저온 유발 유전자들이 시계 유전자에 의해 조절되며, 따라서 식물의 저온에 대한 반응에도 영향을 미칠 가능성이 있다.
CCA1과 LHY 에 더하여 DREB1 조절인자로는 RVE4/LCL1(REVEILLE4/LHY-CCA1-LIKE1)과 RVE8/LCL5가 있으며, 이들은 DREB1 프로모터 영역 중 EE(EVENING ELEMENT)에 결합하여 발현을 조절하는 단백질이다(Fig. 1). 일주기 시계에서 프로모터에 EE가 직접적으로 결합하여 CCA1 및 LHY 에 의해 억제되는 저녁 유전자의 발현을 활성화시킨다[29–31]. 저온 조건에서 RVE4 및 RVE8은 DREB1 프로모터에 직접 결합하여 전사활성화인자로 기능한다. RVE6는 RVE3 및 RVE5와 함께 중복적이지만 조건부로 RVE4 및 RVE8의 활동이 손실된 경우에만 DREB1 발현을 활성화한다. 또한 RVE4 및 RVE8 단백질은 저온 스트레스에 반응하여 세포질에서 핵으로 신속하고 가역적으로 전달되어 DREB1 유전자의 발현을 활성화한다. 스트레스가 없는 정상 조건에서 CCA1 및 LHY 단백질은 DREB1 프로모터에 결합하여 DREB1 발현의 전사 억제자 역할을 하고, 특히 저온 스트레스 조건에서 빠르게 분해되어 DREB1 발현을 유도한다. 그러나 CCA1 및 LHY 단백질이 모두 분해되는 것은 아니며, 저온 스트레스 조건에서 낮은 수준으로 존재하면서 RVE1과 RVE2와 결합하여 저온 유도 유전자 DREB1 발현을 억제한다[32]. CCA1과 LHY는 저온 스트레스 조건에서 RVE4/RVE8와 결합하여 DREB1의 발현을 간접적으로 증가시키면서 COR/RD(COLD-REGULATED/RESPONSIVE TO DEHYDRATION) 유전자의 발현을 직접 조절한다(Fig. 1). 이러한 결과들은 시계 관련 전사인자들이 저온 유발 유전자의 발현을 유도하는 중요한 역할을 하며 이들의 작용 메커니즘에 대한 연구는 식물 생체 시계와 저온 스트레스 반응 사이의 복잡한 관계를 밝힐 뿐만아니라 저온 스트레스에 견딜 수 있는 저항성 작물을 개발할 수 있는 육종 소재를 제공할 것이다.
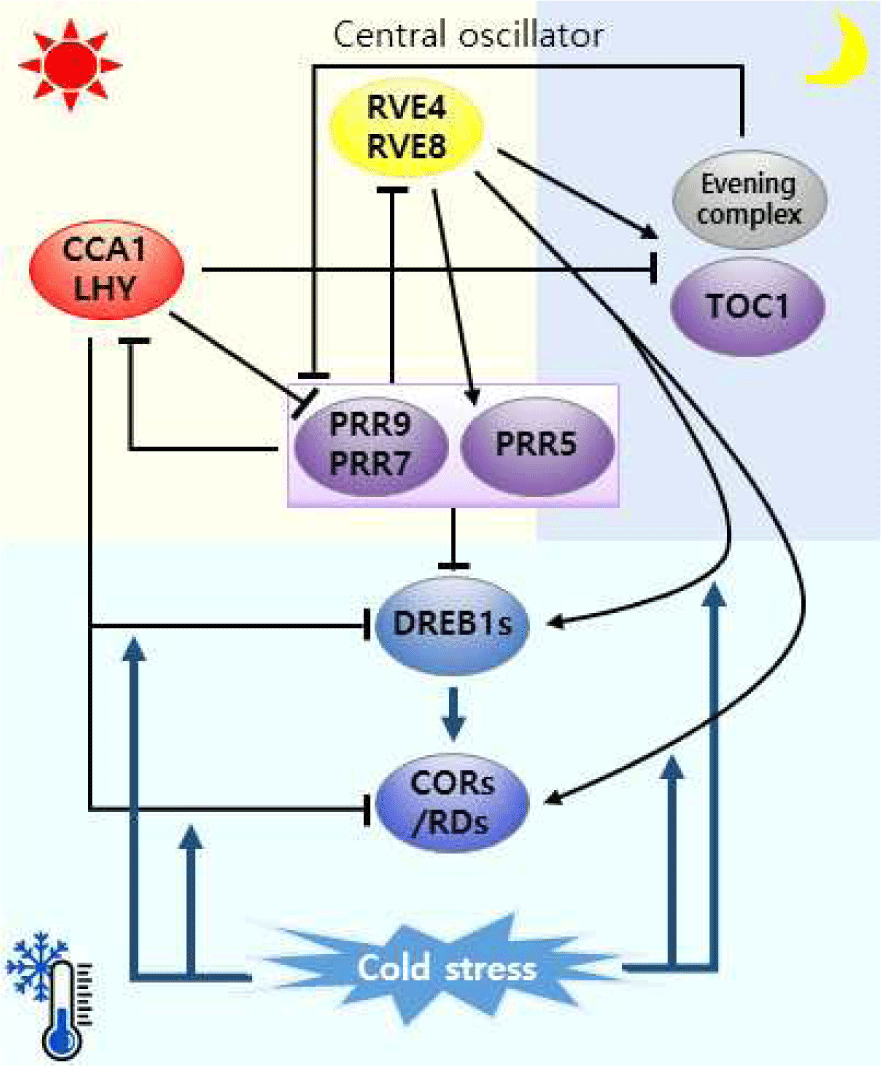
DNA 결합 단백질 LUX와 ELF3 및 ELF4 단백질로 구성된 EC 는 식물 생체시계의 핵심 구성 요소인 전사 억제 복합체이다. EC 는 정상 온도에서 온도 의존적 식물 성장을 조절한다. EC는 4℃에서 더 강한 DNA 결합을 보여주고, 27℃에서 더 약한 결합을 보여줌으로써 온도 센서로 작용할 수 있으며, 이는 ELF4 에 의해 조절된다[33]. 또한 ELF4 는 새싹에서 뿌리로 옮길 수 있으며, 저온은 이러한 움직임을 향상시킨다. ELF3는 발판 단백질이며, 온도 감지 기계의 핵심 구성 요소이다[34]. ELF3의 prion 유사 도메인 내에 포함된 폴리글루타민 반복은 온도 센서로 기능하기 위해 필요하다[35]. 그러나 12℃ 미만의 ELF3에 의한 저온 감지는 아직 입증되지 않았다. 또한 RVE4 및 RVE8의 핵 축적과 CCA1 및 LHY의 분해와 같은 시계 관련 MYB의 번역 후 조절은 저온 스트레스에 대한 반응으로 발생한다[36]. 일주기 구성 요소의 저온 반응 조절의 상세한 메커니즘 분석은 온도 감지 시스템의 해명으로 이어질 수 있다.
자연에서 식물은 다양한 온도 체계 내에서 급격한 온도 변화에 자주 직면하며, 온도 자극에 적응하기 위한 방어 메커니즘을 갖추고 있다. 생육 적온이 22℃인 애기장대의 경우, 실제로는 16℃–28℃까지는 정상적인 생육이 가능하지만, 16℃ 이하나 29℃ 이상의 온도는 스트레스로 인지하여 생장이 심각하게 감소되고, 식물 구조와 형태가 왜곡되어 종자 생산이 감소한다[37]. 애기장대가 생장 기간 동안 고온에 노출되게 되면, 배축 신장, 잎자루 확장, 하배엽 증가 및 작고 얇은 잎을 형성하는데, 이러한 특징을 열형태형성이라 하며, 식물이 뜨거운 토양 표면으로부터 정단분열 조직을 멀리 떨어지게 하고, 잎의 증발 냉각을 촉진하는 데 도움이 된다[38]. 이렇게 스트레스가 없는 주변 온도 적응 전략을 통해 식물은 변동하는 온도 조건에서도 최적의 수준으로 생물학적 활동을 유지할 수 있다[39]. 최근의 연구에서는 고온 스트레스에 대한 식물 반응에도 일주기 시계 유전자가 연관되어 있음이 증명되었다[40,41]. 비록 저온 스트레스 반응과 생체시계와의 관계가 많이 밝혀진 것에 비해 분자 수준에서 고온과의 관계 메커니즘이 잘 밝혀지지는 못했지만, 열스트레스 적응에 관여하는 식물 시계 유전자들에 대한 연구는 현재까지도 진행되고 있다[42–45].
TOC1 및 PRR5, 두 저녁 발현 시계 단백질은 저녁에 고온 반응을 억제하는 역할을 한다고 알려져 있다[42]. TOC1과 PRR5 모두 고온 반응성 하배축 신장을 촉진하는 bHLH (BASIC HELIX-LOOP-HELIX) 전사인자 중 하나인 PIF4 (PHYTOCHROME INTERACTING FACTOR 4 )와 직접 상호작용하여 전사 활성을 억제함으로써 PIF4 매개 성장 촉진을 억제한다[42,46]. 이 기작에서 26S 프로테옴 매개 분해를 위해 TOC1 및 PRR5를 표적으로 하는 F-box 단백질인 ZTL이 관여한다[10,47,48]. ztl 돌연변이 실험을 통하여 PIF4 및 그 표적 유전자 YUC8(YUCCA8)의 발현을 강화하여 고온 반응 하배축 신장에 기여한다는 것이 밝혀졌다[49,50]. ztl 돌연변이에서 PIF4 발현은 고온에 의해 증가했지만 PIF4의 기본 수준은 야생형 애기장대 식물의 수준보다 낮았는데, 이러한 결과는 ZTL이 PIF4 전사뿐만 아니라, PIF4 활성에도 영향을 미친다는 것을 설명한다. 애기장대 ztl 돌연변이 실험에서 야생형 식물의 하배축 신장은 고온(28℃)에 의해 4배 이상 촉진되었으나, ztl 돌연변이의 하배축 신장은 고온에 의해 2배 증가하였고, ZTL 과발현 식물은 고온 조건에서 야생형보다 더 길어졌다. 이러한 결과는 ZTL이 열형태형성에 관여하여 고온에서 하배축을 신장시키는 역할을 한다는 것을 말해준다. ZTL은 TOC1 및 PRR5를 분해하는 E3 lligase로, TOC1 및 PRR5의 발현 수준을 낮추고 PIF4를 증가시켜 열형태형성을 촉진한다. 따라서 TOC1과 PRR5 발현 수준이 증가하는 저녁 시간에는 PIF4 매개 온도 반응 성장을 억제한다. 또한 PIF4는 EC에 의해 발현이 억제되며, 고온 반응 하배축 신장 특성을 보인다[51,52]. EC 의 핵심 구성 요소인 ELF3의 발현은 야생형보다 ztl 돌연변이에서 더 높았는데, 이는 ZTL 유전자가 ELF3 단백질 발현량을 조절하는 것으로 보인다. ztl 돌연변이에서 고온에 대한 PIF4 및 그 표적 유전자의 발현은 TOC1‐OX 및 PRR5‐OX 식물과 유사했고[42], ztl 돌연변이의 유전자 발현 및 하배축 신장에서의 온도 조절 결함은 오히려 ztl;toc1;prr5 삼중 돌연변이에서 크게 회복되었다. 결과적으로 ZTL은 TOC1 및 PRR5, 그리고 EC 등을 통해 PIF4 발현을 긍정적으로 조절하여 고온 반응성 하배축 신장을 향상시킨다(Fig. 2). 또한 PRR5는 고온 매개 하배축 신장을 억제하는 것으로 알려져 있다. PRR5 단백질은 야생형 및 ztl 돌연변이 모두에서 고온에 의해 유사하게 증가하기 때문에 ZTL-PRR5 모듈은 PIF4의 최대 활성에 영향을 미침으로써 고온에 대한 하배축 신장의 민감도를 결정한다[42,45].
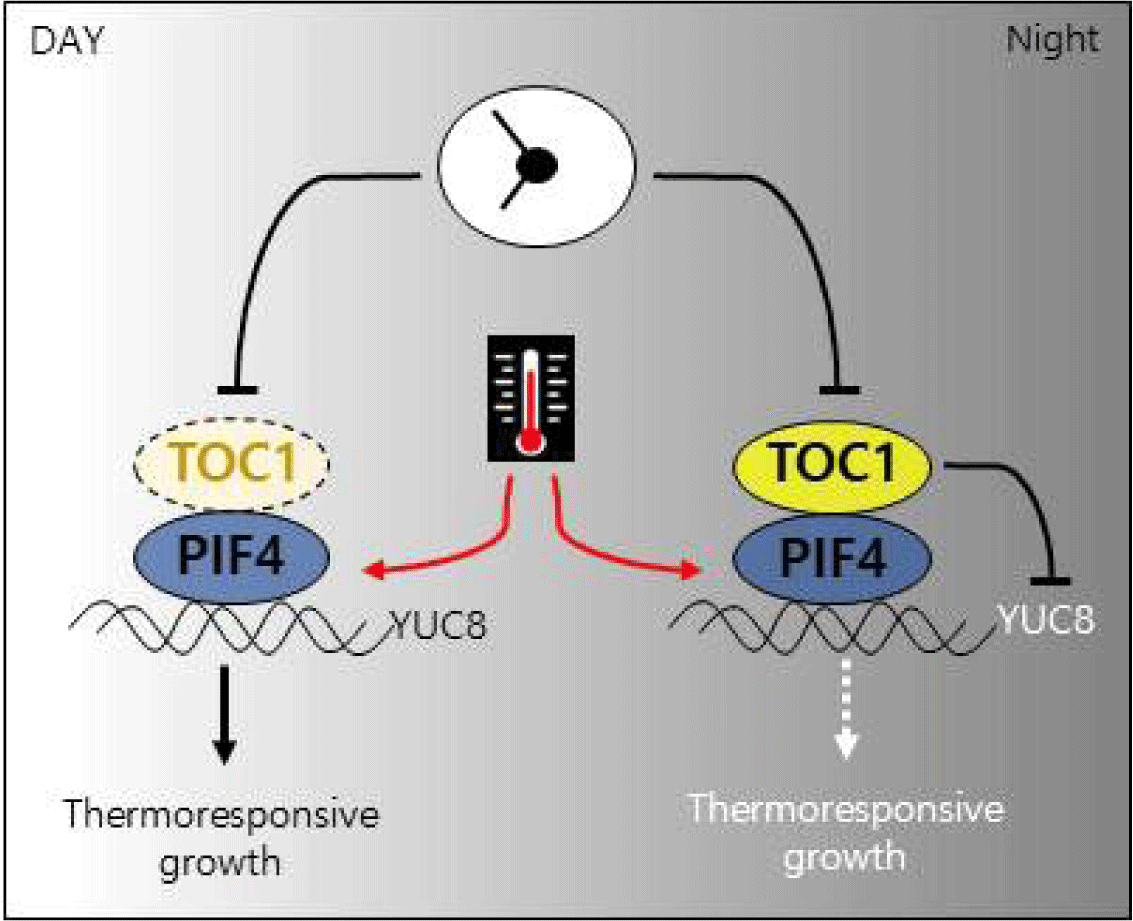
ZTL은 열형태형성을 조절하는 것 외에도 단백질 발현 시스템을 통해 내열성을 증가시킨다[6]. 고온에서 ZTL 발현의 증가는 고온 스트레스 하에서 식물의 생존을 촉진할 수 있다. 열형태형성과 달리 ztl 돌연변이의 내열성 결함은 TOC1 및 PRR5 돌연변이에 의해 복원되지 않기 때문에 ZTL은 별개의 메커니즘을 통해 두 가지 고온 반응을 조절하는 것으로 보인다[53]. 또한 HSP90 클라이언트 단백질인 ZTL은 폴리유비퀴틴화 매개 단백질 분해를 통해 고온 유도 단백질 응집을 해소하여 내열성을 개선한다[6]. HSP90은 옥신 수용체 TIR1(TRANSFORT INHIBITOR RESPONSE 1)을 안정화시켜 열형태형성을 긍정적으로 제어하는것으로 밝혀졌다[54]. HSP90은 GI와 관련이 있고, 밤에 TOC1과 PRR5의 분해에 필요한 ZTL의 성숙을 조절한다. 종합하면, HSP90은 ZTL-TOC1/PRR5 신호 모듈과 TIR1 옥신 수용체를 통해 열형태형성을 조절한다.
정상 온도 범위에서 온도 변화를 수용하여 식물이 항상성을 유지하게 하거나, 극단적인 저온과 고온에서 식물의 생존과 생장 유지를 위해 세포 내 단백질의 성질을 변화시키고, 형태형성의 변화를 유도하는데 있어 생체시계 유전자는 중요한 역할을 하며, 특히 중앙진동자에 연관된 시계 유전자인 TOC1이 관여한다는 연구 결과는 매우 흥미롭다. 나아가 식물의 전체 생체리듬을 유지하는 유전자들이 저온과 고온 스트레스에 대한 식물의 반응 메커니즘에 관여한다는 연구 결과는 지구 온난화에 대비하여 온도 스트레스 내성이 향상된 작물을 육성하는데 있어 중요한 육종 소재가 될 수 있으며, 미래 식물 자원 확보와 식량 안정성을 위해 기여할 수 있다.
요 약
식물의 생장과 발달은 주변 온도 조건에 의해 광범위하게 영향을 받는다. 극한의 기온은 전 세계적으로 광범위한 농작물 손실을 초래하고, 농업 목적으로 사용할 수 있는 토지의 양에 심각한 제한을 가한다. 따라서 다른 비생물학적 스트레스와 함께 온도 스트레스에 대한 내성을 가진 작물의 개발이 시급하다. 식물은 일장과 함께 하루 중에 변화하는 온도 신호를 기억하여 생체리듬을 만들고, 이는 생리적 과정 및 대사 과정을 시기적절하게 조절하게 한다. 하지만, 동시에 온도 변화에 둔감하게 적응하면서 생체리듬을 보존하여 생육과 생존을 유지할 수 있다(온도보상). 식물 생체시계 유전자가 식물의 온도에 대한 반응을 조절한다는 최근 연구 결과가 보고되면서 생체시계 유전자를 통하여 작물의 온도 스트레스에 대한 내성을 획득하려는 시도가 더 많은 관심을 끌고 있다. 예를 들면, DREB1/CBF 전사인자는 저온 스트레스 내성 획득을 위한 전사 조절 네트워크에서 마스터 조절 인자로 알려져 있다. 또한 식물은 열형태형성을 통해 고온 스트레스에 적응하면, 이때 PIF4 전사인자가 관여한다. 하루 동안에도 DREB1 및 PIF4 발현은 시간에 따라 변화하며, 저온과 고온에 대한 식물의 반응도 일일주기를 보인다. 최근 연구는, 저온 신호전달 전사인자들로 구성된 DREB1 경로에 중앙진동자 유전자인 CCA1/LHY 와 PRR5/7/9 그리고 저녁인자(EC )로 알려진 REV4/REV8 등이 관여하며, 열형태형성 조절에도 또다른 중앙진동자인 TOC1과 조절 단백질 ZTL이 관여하는 것을 제시하였다. 본 리뷰에서는 식물의 온도 변화 반응과 연관된 시계 유전자들을 소개하고자 하며, 이는 급변하는 지구의 기후에 식물이 살아남을 수 있는 전략을 세우는데 이용될 수 있을 것이다.






